7 avril 2020
Genci
La calcul intensif au service de la connaissance
En charge de mettre à disposition des moyens de calcul et de traitement de données massives performants, GENCI a pour mission, au niveau national et européen, de favoriser l’usage du calcul intensif associé à l’Intelligence Artificielle au bénéfice des communautés de recherche académique et industrielle.
L'équipe Bioinformatique structurale du laboratoire GBCM
Le laboratoire GBCM, Génomique, bioinformatique et chimie moléculaire, compte trois équipe de recherche. L’équipe de bioinformatique structurale, modélisation moléculaire et drug design (M2D2) est spécialisée dans l’analyse des interactions protéine-protéine ou protéine-petite molécule.
Depuis sa création, l’équipe développe et utilise des méthodes de modélisation moléculaire et de criblage in silico et les applique à l’identification de molécules à visée thérapeutique pour cibler les interactions protéine-protéine. Elle a, depuis, étendu son champ d’activités. L’équipe M2D2 a développé de nombreuses collaborations, internes au Cnam avec les équipes ILJ et MSDMA du Cedric et le laboratoire Satie et nationales avec notamment des équipes Inserm de l’institut Imagine et de Paris Descartes, de l’université de Nice Sophia Antipolis/CNRS, le LCT de Sorbonne Universités/CNRS et le Liris de l’Insa/CNRS/Lyon. Son savoir-faire repose sur différentes méthodes de modélisation moléculaire des interactions protéine-protéine et protéine-petite molécule, des méthodes de criblage structure ou ligand-based et de méthodes de représentation et de visualisation de la structure et des propriétés des protéines.
Clin d'oeil au Cray-2 conservé au musée des Arts et Métiers, supercalculateur utilisé dans les années 80!
Issu du supercalculateur Cray-1 conçu en 1976 par l’ingénieur américain Seymour Cray, le Cray-2 est au moment de sa sortie l’ordinateur le plus puissant au monde, le second à dépasser la barre du gigaflop (un milliard d’opérations par seconde), un an après le M-13 russe.
Il met en œuvre le principe du calcul vectoriel, selon lequel une seule instruction provoque une cascade de calculs effectués simultanément par plusieurs processeurs. Il dispose d’une architecture très compacte en forme de C pour diminuer les distances entre composants et augmenter la vitesse de calcul. Pour dissiper la chaleur produite par ses centaines de milliers de puces, l’ensemble baigne dans un liquide conducteur de chaleur et isolant, refroidi par de l’eau. Le Cray-2 était l’outil idéal des gros centres de calcul scientifique pour les domaines de la météorologie ou de la dynamique des fluides. Cet exemplaire a été utilisé à l’École polytechnique de 1985 à 1993. Lionel Dufaux, responsable des collections Énergie et Transports au musée des Arts et Métiers. 
















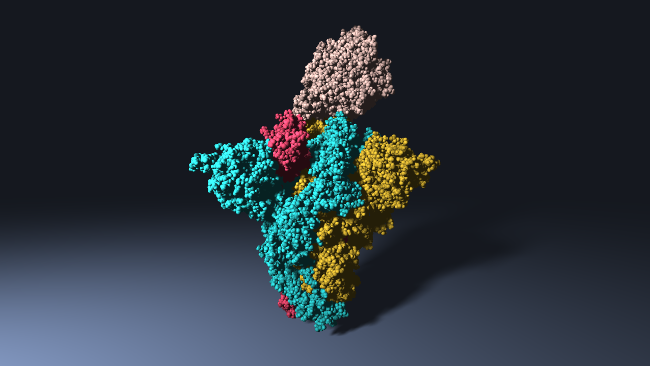
 En mars 2020, Sorbonne Université à Paris s'allie à plusieurs chercheurs en France et dans le monde pour étudier la structure moléculaire du COVID19 en collaboration avec l'Institut Pasteur, un institut leader mondial dans le domaine des maladies infectieuses émergentes.
En mars 2020, Sorbonne Université à Paris s'allie à plusieurs chercheurs en France et dans le monde pour étudier la structure moléculaire du COVID19 en collaboration avec l'Institut Pasteur, un institut leader mondial dans le domaine des maladies infectieuses émergentes.